class: center, middle, inverse, title-slide # Algoritmos Bayesianos para clasificación y regresión ## Curso de aprendizaje automático para el INE ### Víctor Gallego y Roi Naveiro ### 2019-05-27 --- ## ¿Por qué usar el enfoque Bayesiano? * Previene el **overfitting** * Aporta métodos automáticos para determinar la complejidad de los modelos, basados en datos. * Permite **cuantificar la incertidumbre**, es decir, no solo nos da una predicción puntual, si no que podemos obtener un intervalo predictivo para saber cómo de confiado está el modelo. --- class: middle, center, inverse # Regresión Bayesiana --- ## Modelo de Regresión * Objetivo: predecir uno o más targets **contínuos** `\(t\)` a partir de un vector D-dimensional de inputs `\(x\)`. * Modelo de ruido normal, `\(t = y(x,w) + \epsilon\)`, donde `\(y(x,w) = \sum_{j=0}^{M-1} w_j \phi_j(x) = w^\top \phi(x)\)`. `\begin{equation} p(t \vert x, w, \beta) = \mathcal{N}(t \vert y(x,w), \beta^{-1}) \end{equation}` -- * Si tenemos datos `\(x_1, \dots, x_N\)`, `\(t_1, \dots, t_N\)` y asumimos que son muestras iid de `\(p(t \vert x, w, \beta)\)`, la verosimilitud es `\begin{equation} p(\boldsymbol{t} \vert X, w) = \prod_{n=1}^N \mathcal{N}(t_n \vert w^\top \phi(x_n), \beta^{-1}) \end{equation}` * Donde `\(\boldsymbol{t}\)` es el vector de los `\(N\)` targets y `\(X\)` la matriz de datos. * Maximizando la log-verosimilitud con respecto a `\(w\)` obtenemos la solución de mínimos cuadrados. --- ## Enfoque Bayesiano (1) * Tratamos los parámetros desconocidos como variables aleatorias. * Empezaremos asumiendo que conocemos la precisión `\(\beta\)`. * Ponemos distribuciones a priori sobre los pesos `\(w\)`. * Como la verosimilitud es la exponencial de una función cuadrática de w, el prior conjugado es normal `\begin{equation} p(w) = \mathcal{N}(w \vert m_0, S_0) \end{equation}` --- ## Enfoque Bayesiano (2) * Podemos calcular la distribución a posteriori `\begin{equation} p(w \vert \boldsymbol{t}, X) \propto p(t \vert X, w)p(w) \end{equation}` * Gracias a la conjugación, la distribución a posteriori será también Gaussiana. `\begin{equation} p(w \vert \boldsymbol{t}, X) = \mathcal{N}(w \vert m_N, S_N) \end{equation}` * Donde `\begin{eqnarray*} m_N &=& S_N(S_0^{-1} m_0 + \beta \Phi^\top \boldsymbol{t}) \\ S_N^{-1} &=& S_0^{-1} + \beta \Phi^\top \Phi \end{eqnarray*}` * `\(\Phi\)` es la matriz de diseño. La final i-ésima es el vector `\([\phi_0(x_i), \dots, \phi_{M-1}(x_i)]\)`. -- * **Ojo**: no siempre podremos calcular analíticamente la distribución a posteriori... --- ## Enfoque Bayesiano (3) * **Ejercicio**: demostrar que si consideramos una distribución a priori infinitamente ancha, `\((S_0 = \alpha^{-1} I, \alpha \rightarrow 0)\)`, la media (moda) de la distribución a posteriori converge a la solución de máxima verosimilitud dada por `\begin{equation} w_{ML} = (\Phi^\top \Phi)^{-1} \Phi^\top \boldsymbol{t} \end{equation}` --- ## Enfoque Bayesiano (4) * Consideremos el caso de distribución a priori isotrópica de media 0. `\begin{equation} p(w \vert \alpha) = \mathcal{N}(w \vert 0, \alpha^{-1} I) \end{equation}` -- <center> 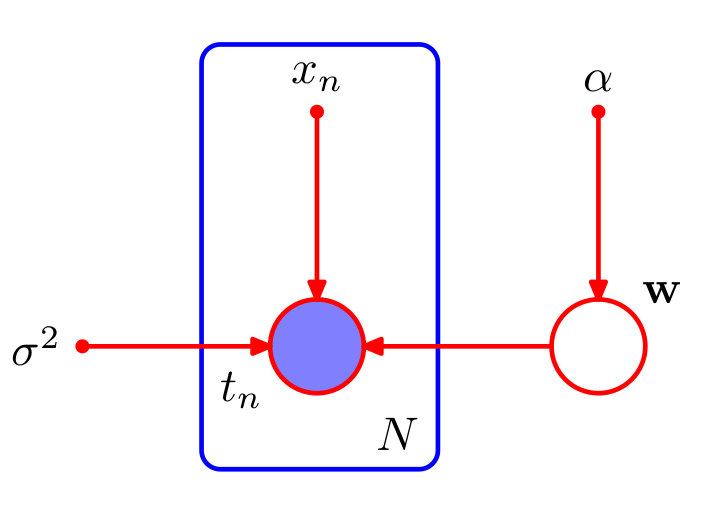 </center> --- ## Enfoque Bayesiano (4) * **Ejercicio:** demostrar que maximizar el log-posterior con respecto a `\(w\)`, equivale a encontrar el estimador de máxima verosimilitud de los pesos del problema de regresión con regularización `\(L2\)`. -- * En el caso bajo consideración, podemos escribir el log-posterior como la suma del log-prior y la log-verosimilitud. `\begin{equation} -\frac{\beta}{2} \sum_{n=1}^N \left \lbrace t_n - w^\top \phi(x_n)\right \rbrace^2 - \frac{\alpha}{2} w^\top w + \text{cte} \end{equation}` * Maximizar este posterior es equivalente a encontrar la solución de regresión ridge con parámetro de regularización `\(\alpha/\beta\)`. --- ## Enfoque Bayesiano (5) * En la práctica, estamos interesados en hacer predicciones del target `\(t\)` asociado a un nuevo input `\(x\)`. * Esto requiere evaluar la **distribución predictiva a posteriori** `\begin{equation} p(t \vert \boldsymbol{t},\alpha, \beta) = \int p(t \vert w, \beta)p(w \vert \boldsymbol{t}, \alpha, \beta) dw \end{equation}` -- * Es fácil probrar que `\(p(t \vert \boldsymbol{t},\alpha, \beta) = \mathcal{N}(t \vert m_N^\top \phi(x), \sigma_N^2(x))\)`, con `\begin{equation} \sigma_N^2(x) = \frac{1}{\beta} + \phi(x)^\top S_N \phi(x) \end{equation}` * Dos fuentes de incertidumbre: la asociada al modelo y la asociada al desconocimiento de los `\(w\)` (en el límite de muchos datos esta última es cero). -- * **Ojo**: la distribución predictiva a posteriori no siempre puede calcularse analíticamente... --- ## Enfoque Bayesiano (6) * En todos los casos hemos asumido conocido el valor de la precisión `\(\beta\)`. * Si fuese desconocido, deberíamos poner un prior sobre `\(w, \beta\)`. * Eligiendo una distribución Gaussiana-Gamma, se mantiene la conjugación. * En este caso la distribución predictiva es una t de Student. * Si también queremos asignar un prior al parámetro `\(\alpha\)` (hiperprior), debemos recurrir a técnicas de inferencia aproximada. --- class: middle, center, inverse # Clasificación Bayesiana --- ## Regresión Logística Bayesiana (1) * Más complicado que el caso de regresión debido a la no-linealidad de la sigmoide. * Debemos utilizar algún tipo de aproximación del posterior: **aproximación de Laplace**. -- * Queremos aproximar cierta distribución `\(p(z) = \frac{1}{Z} f(z)\)`. * Aproximamos mediante una Normal `\(q(z)\)` centrada en la moda de `\(p(z)\)`: buscamos `\(z_0\)` tal que `\(\nabla_z f(z) |_{z=z_0} = 0\)`. * Desarrollamos en Taylor hasta orden 2 en `\(z_0\)`: `\(\log f(z) \approx \log f(z_0) - \frac{1}{2} A (z - z_0)^2\)`, donde `\(A = - \nabla_z \nabla_z \log f(z) |_{z=z_0}\)`. * Tomando exponenciales `\(f(z) \approx f(z_0) \exp \lbrace -\frac{A}{2} (z - z_0)^2 \rbrace\)` * Normalizando, `\(q(z) = ( \frac{A}{2\pi})^{1/2} \exp \lbrace -\frac{A}{2} (z - z_0)^2 \rbrace\)` resultando una Normal. --- ## Ejemplo de Aproximación de Laplace <center> 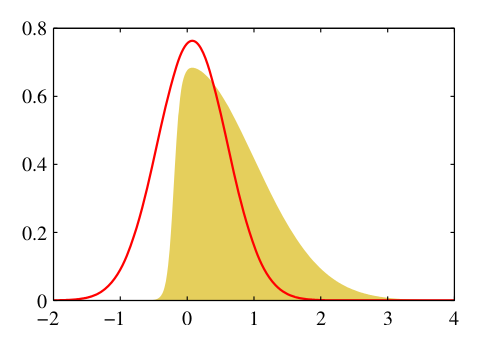 </center> * **Problemas** de la aproximación: * Solo se puede aplicar directamente a **variables reales** (si no, hay que reparametrizar). * Falla al describir propiedades globales de la distribución aproximada. --- ## Regresión Logística Bayesiana (2) * Particularizándolo al caso de regresión logística, * El prior es Gaussiano: `\(p(w) = \mathcal{N}(w| m_0, S_0)\)`. * Para el posterior, `\(p(w|t) \propto p(w) p(t|w)\)`, y tomando logaritmos queda `\begin{equation} \log p(w|t) = - \frac{1}{2}(w - m_0)^\intercal S_0^{-1} (w - m_0) + \sum_{n=1}^N \lbrace t_n \log y_n + (1 - t_n)\log(1 - y_n) \rbrace + C \end{equation}` * donde `\(y_n = \sigma (w^\intercal \phi_n)\)` -- * Para usar Laplace, obtenemos el punto `\(w_{MAP}\)`, que será la media de la Gaussiana. * La precisión será `\(S_N = - \nabla \nabla \log p(w|t) = S_0^{-1} + \sum_{n=1}^N y_n (1 - y_n) \phi_n \phi_n^\intercal\)` * Con lo que `\(p(w|t) \approx q(w) = \mathcal{N}(w | w_{MAP}, S_N)\)`. --- ## Distribución predictiva * Dado un nuevo input `\(\phi = \phi(x)\)`, queremos predecir su clase: `\begin{equation} p(C_1 | \phi, t) = \int p(C_1 | \phi, w) p(w | t) dw \approx \int \sigma (w^\intercal \phi) q(w) dw \end{equation}` -- * Esta integral no es analítica, luego tenemos dos opciones: * **Aproximación por MC**: `\(p(C_1 | \phi, t) \approx \frac{1}{K} \sum_{k=1}^K p(C_1 | \phi, w^{(k)}), \qquad w^{(k)} \sim q(w)\)`. -- * **Aproximación analítica**: en lugar de sigmoide usamos la función probit, ya que `\(\sigma(a) \approx \Phi(\lambda a)\)` y su convolución respecto a la Gaussiana es analítica (otra probit) `\begin{equation} \int \Phi (\lambda a) \mathcal{N}(a | \mu, \sigma) da = \Phi \left( \frac{\mu}{(\lambda^{-2} + \sigma^2)^{1/2}} \right) \end{equation}` * Tras unos ajustes, queda que `\begin{equation} p(C_1 | \phi, t) \approx \sigma (\kappa ( \sigma^2) \mu) \end{equation}` * donde `\(\mu = w_{MAP}^\intercal \phi\)`, `\(\sigma = \phi^\intercal S_N \phi\)` y `\(\kappa(\sigma^2) = (1 + \pi \sigma^2 / 8)^{-1/2}\)`. --- class: middle, center, inverse # Inferencia Aproximada --- ## Introducción * Un tarea central de la estadística Bayesiana es encontrar la distribución a posteriori `\(p(Z \vert X)\)`, así como valores esperados respecto a esta distribución. * `\(Z\)` son variables latentes y `\(X\)` variables observadas. * En muchos modelos, esta tarea es **inviable**: 1. **Variables contínuas**: las integrales resultantes no tienen solución analítica. La alta dimensionalidad del espacio no permite el uso de muchas técnicas numéricas. 2. **Variables discretas**: marginalizar requiere sumar sobre todas las posibles combinaciones de variables ocultas, que pueden crecer exponencialmente. --- ## Introducción * En estos casos debemos recurrir a **inferencia aproximada** * Dos clases de inferencia aproximada. 1. Técnicas **estocásticas** (MCMC). Asintóticamente **exactas**. La aproximación viene de no tener tiempo computacional infinito. **Costosas computacionalmente.** 2. Técnicas **deterministas** (VI). Aproximaciones analíticas a la distribución a posteriori. **Nunca** son exactas. **Eficientes**. --- class: middle, center, inverse # Markov Chain Monte Carlo --- ## Repaso de Monte Carlo * El objetivo de los métodos Monte Carlo es el de calcular **esperanzas** con respecto a cierta distribución `\(p(z)\)` `\begin{equation} \mathbb{E} \left[ f(z) \right] = \int_{\mathcal{Z}} f(z)p(z)dz \end{equation}` * **Idea**: obtenemos una **muestra finita** `\(z^{(l)}, l=1,\ldots,L\)` de `\(p(z)\)`, por lo que podemos aproximar mediante `\begin{equation} \mathbb{E} \left[ f(z) \right] \approx \frac{1}{L} \sum_{l=1}^L f(z^{(l)}) \end{equation}` * ¿Qué hacer cuando no podemos muestrear directamente de `\(p(z)\)`? -- * Muestreo por rechazo (**rejection sampling**): no es muy general. * Muestreo por importancia (**importance sampling**): solo para calcular integrales, no permite obtener muestras directamente: con lo que si queremos cambiar la `\(f(z)\)`, hay que repetir todo desde 0 (costoso). * **Markov Chain Monte Carlo (MCMC)**: general y obtiene muestras directamente. --- ## MCMC: fundamentos * **Objetivo**: obtener muestras de `\(p(z)\)`. * **Asumimos**: sabemos evaluar `\(p(z)\)` salvo constante de proporcionalidad. * Es decir, basta con saber evaluar `\(\tilde{p}(z) = Z p(z)\)`. * **Idea**: generar muestras de una cadena de Markov cuya distribución invariante (límite) sea `\(p(z)\)`. -- * **Esquema general**: 1. A partir de la muestra actual `\(z^{(\tau)}\)`, generar una muestra *candidata* mediane una distribución (**proposal**), `\(z^* \sim q(z | z^{(\tau)})\)`. 2. Aceptamos la candidate mediante algún criterio. 3. Si es aceptada, `\(z^{(\tau + 1)} = z^*\)`. Si no, `\(z^{(\tau + 1)} = z^{(\tau)}\)`, e iteramos. * Las muestras `\(z^{(1)}, z^{(2)}, \ldots\)` forman una cadena de Markov. --- ## Algoritmo de Metropolis * El proposal tiene que ser simétrico: `\(q(z_A|z_B) = q(z_B | z_A)\)`. * Aceptamos la muestra con probabilidad `\begin{equation} A(z^*, z^{(\tau)}) = \min (1, \frac{\tilde{p}(z^*)}{\tilde{p}(z^{(\tau)})}) \end{equation}` -- * Típicamente, `\(q(z | z^{(\tau)}) \sim \mathcal{N}(z | z^{(\tau)}, \sigma)\)` (Random Walk Metropolis) * Visualización interactiva en https://chi-feng.github.io/mcmc-demo/app.html#RandomWalkMH,banana. --- ## ¿Por qué funciona Metropolis? * Un proceso estocástico `\(z^{(1)}, z^{(2)}, \ldots\)` es una **cadena de Markov** si verifica `\begin{equation} p(z^{(m+1)} | z^{(m)}, z^{(m-1)}, \ldots z^{(1)}) = p(z^{(m+1)} | z^{(m)}) \end{equation}` * Una cadena de Markov homogénea viene especificada por la distribución inicial y las **probabilidades de transición** ( `\(T\)` no cambia con `\(m\)` ): `\begin{equation} T(z^{(m)}, z^{(m+1)}) = p(z^{(m+1)} | z^{(m)}) \end{equation}` * Una distribución `\(p^*(z)\)` queda invariante bajo la cadena si `\begin{equation} p^*(z) = \sum_{z'} T(z', z) p^*(z') \end{equation}` -- * Una condición suficiente para que `\(p^*(z)\)` sea invariante es **eligiendo** `\(T\)` de forma que satisfaga la condición de **balance detallado** `\begin{equation} p^*(z)T(z, z') = T(z', z)p^*(z') \end{equation}` --- ## ¿Por qué funciona Metropolis? * Ahora bien, tomamos ( `\(z\)` es la última muestra y `\(z'\)` es la muestra propuesta ) `\begin{equation} T(z, z') = p(z' | z) = A(z', z) q(z' | z) \end{equation}` * por lo que de imponer la condición, queda que `\begin{equation} \frac{A(z', z)}{A(z, z')} = \frac{p(z')}{p(z)} \frac{q(z|z')}{q(z'|z)} \end{equation}` donde el último factor desaparece (pues era **simétrico**), y una tasa de aceptación que satisface lo anterior es `\begin{equation} A(z',z) = \min (1, \frac{p(z')}{p(z)}) \end{equation}` --- ### Algoritmo de Metropolis-Hastings * Generalización que permite el uso de **cualquier proposal**. * Ahora aceptaremos una muestra con probabilidad: `\begin{equation} A(z^*, z^{(\tau)}) = \min (1, \frac{p(z^*) q(z^{(\tau)} | z^*) }{p(z^{(\tau)}) q(z^* | z^{(\tau)}) }) \end{equation}` -- ### Efecto de hiperparámetros * Si el proposal es `\(q(z | z^{(\tau)}) \sim \mathcal{N}(z | z^{(\tau)}, \rho)\)`, valores **bajos** de `\(\rho\)` hacen que la tasa de aceptación sea alta, pero explore muy lentamente. Valores **altos** de `\(\rho\)` provocan que se explore más regiones del espacio, a costa de aumentar las muestras rechazadas. <center> 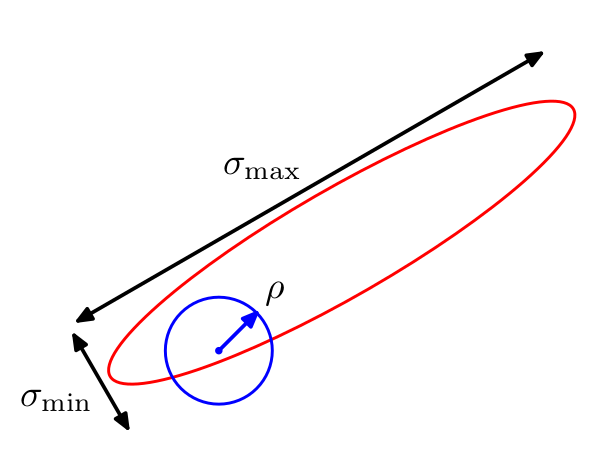 </center> --- ## Gibbs sampling (1) * Caso especial de Metropolis-Hastings, utilizado cuando * El objetivo es multidimensional: `\(p(z) = p(z_1, \ldots, z_M)\)`. * Se conocen las marginales condicionadas `\(p(z_i | z_{\setminus i})\)`. * **Esquema general**: iremos iterando muestras de cada una de las distribuciones condicionales anteriores. --- ## Gibbs sampling (2) * **Ejemplo**: supongamos que la distribución objetivo es `\(p(z) = p(z_1, z_2, z_3)\)`. * En la iteración `\(i\)`, hemos obtenido por muestreo valores `\(z_1^{(i)}, z_2^{(i)}, z_3^{(i)}\)`. * Obtenemos nuevas muestras `\(z_1^{(i+1)}, z_2^{(i+1)}, z_3^{(i+1)}\)` mediante: * `\(z_1^{(i+1)} \sim p(z_1 | z_2^{(i)}, z_3^{(i)})\)`. * `\(z_2^{(i+1)} \sim p(z_2 | z_1^{(i+1)}, z_3^{(i)})\)`. * `\(z_3^{(i+1)} \sim p(z_3 | z_1^{(i+1)}, z_2^{(i+1)})\)`. -- * Si en lugar de muestrea tomamos la moda de cada condicional, obtenemos el algoritmo de **modas condicionadas iteradas (ICM)**. --- ## Gibbs sampling (3) * **Ejemplo** : <span style="color:red">Gaussiana bidimensional</span>, e <span style="color:blue">iteraciones del Gibbs sampler</span> <center> 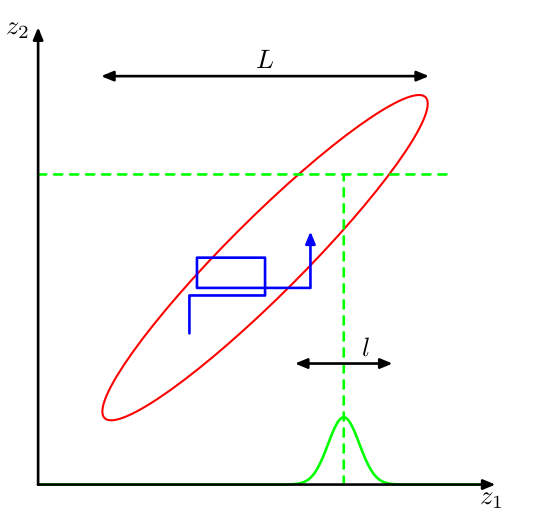 </center> -- * **Problema**: avance **lento** si hay muchas correlaciones entre variables. --- ## Hamiltonian Monte Carlo (HMC) * ¿Podemos mejorar el camino aleatorio de la cadena? * ¿Además de aliviar el problema del tamaño del paso en Metropolis-Hastings? -- * **Intuición**: hasta ahora solo hemos utilizado `\(p(z)\)` (o condicionales suyas). ¿Por qué no utilizar también la información del gradiente `\(\nabla \log p(z)\)`? * También conocido como Hybrid Monte Carlo, HMC es adecuado para **espacios continuos**: * Permite dar grandes saltos en el espacio. * Baja tasa de muestras rechazadas. * Necesita evaluar el gradiente de la logprobabilidad respecto a `\(z\)`. * **Se espera que la mejor exploración (mayor muestra efectiva) compense el coste computacional de evaluar el gradiente**. --- ## Hamiltonian Monte Carlo (2) * El objetivo es obtener muestras de `\(p(z) = \frac{1}{Z} \exp \lbrace -E(z) \rbrace\)`. * `\(E(z)\)` se interpreta como **energía potencial** en el estado `\(z\)`. * ¿Qué ocurre si sólo imponemos la siguiente dinámica (**descenso por el gradiente**)? `\begin{equation} z^{(t+1)} = z^{(t)} - \eta \nabla_z E(z^{(t)}) \end{equation}` -- * Nos quedariamos en el MAP. No basta solo con eso, hay que añadir estocasticidad para que explore toda la región `\(p(z)\)` * Para ello, añadimos un término de **energía cinética**, añadiendo **variables auxiliares de momento** `\(r\)`. `\begin{equation} p(z,r) = \frac{1}{Z} \exp \lbrace -E(z) - K(r) \rbrace = \frac{1}{Z} \exp \lbrace -H(z,r) \rbrace \end{equation}` * donde `\(K(r) = \frac{1}{2} r^\intercal r\)` (ruido Gaussiano). --- ## Hamiltonian Monte Carlo (3) * La dinámica del Hamiltoniano preserva la cantidad `\(H(z,r)\)` a lo largo de las trayectorias, luego también dejará invariante la distribución `\(p(z,r)\)`. `\begin{align} \frac{dz}{dt} &= +\nabla_r H \\ \frac{dr}{dt} &= -\nabla_z H \end{align}` * Es delicado discretizar numéricamente la anterior ODE, hay que tener en cuenta errores numéricos: * Se corrigen aceptando bajo probabilidad `\begin{equation} \min (1, \exp \lbrace H(z,r) - H(z', r') \rbrace) \end{equation}` * Conviene ir remuestreando el momeno `\(r\)` cada pocas iteraciones. * Ejemplo de visualización: https://chi-feng.github.io/mcmc-demo/app.html#HamiltonianMC,donut * Versión adaptativa (tamaño del paso) mucho más eficiente: **No U-Turn Sampler** https://arxiv.org/abs/1111.4246 --- ## Convergencia (1) <center>  </center> * Conviene utilizar varias cadenas partiendo de condiciones iniciales aleatorias: * Izquierda: 50 iteraciones. * Centro: 1000 iteraciones. * Derecha: muestras tras descartar la primera mitad de cada cadena (**burn-in**). * También se puede tomar 1 muestra cada `\(n > 1\)` iteraciones para reducir autocorrelaciones (**thining**). --- ## Convergencia (2) Algunos test para diagnosticar la convergencia y el rendimiento son: * Estadístico `\(\hat{R}\)` (Potencial Scale Reduction) * Sumariza correlaciones intra-cadena y entre cadenas, para una variable del modelo. * `\(\hat{R} >> 1\)`: no hay convergencia. * `\(\hat{R} \approx 1\)`: no la garantiza. * Tamaño de muestra efectiva `\(N_{eff}\)`: * Corrige el número total de muestras para tener en cuenta la autocorrelación. * Caso peor: `\(z^{(1)} = z^{(2)} = \ldots = z^{(N)}\)`, `\(N_{eff} = 1\)`. * La precisión de la estimación será proporcional a `\(\frac{1}{\sqrt{N_{eff}}}\)`. --- class: middle, center, inverse # Extra: breve intro a Stan --- ## Stan como PPL * Lenguajes de **programación probabilística** (PPL): extensión de un lenguaje de programación normal con nuevas operaciones como **sample** ( `\(\sim\)` ) y **condicionar**. * Separación entre **modelo** e **inferencia**, permitiendo automatizar el siguiente ciclo (propuesto por Box): <center>  </center> * Mucho desarollo en los últimos años, aunque todavía experimentales * En R, el más popular y estable es **rStan**: https://mc-stan.org/users/interfaces/rstan --- ## Flujo de trabajo con un PPL 1. Escribir el **modelo probabilístico** (priores + verosimilitudes). 2. Ejecutar el **motor de inferencia** (MCMC, VI, MAP, ...). 3. Diagnosticar la **convergencia**. 4. Realizar la **inferencia** (muestras del posterior). --- ## Modelos en Stan * Se escriben en un mini-lenguaje (DSL) simple (no en R) para que posteriormente puedan ser compilados a C. * Especificamos **parámetros** y **variables latentes**. <center>  </center> --- ## Motores de inferencia en Stan * HMC/NUTS <center>  </center> * VI (**V**ariational **B**ayes, siguiente capítulo) <center>  </center> --- ## Diagnósticos en Stan <center>  </center> * También visualizaciones: * plot(fit) * pairs(fit, pars = c("mu", "tau")) * traceplot(fit, pars = c("mu", "tau"), inc_warmup = TRUE, nrow = 2) --- class: middle, center, inverse # Inferencia Variacional --- ## Introducción * VI convierte la inferencia en un problema de optimización. * Busca aproximaciones **analíticas** a la distribución a posteriori. * Basado en *cálculo de variaciones*. * Optimización de funcionales: encontrar funciones que maximicen o minimicen el funcional. * En principio, el cálculo de variaciones es exacto. La aproximación nace de restringir el espacio de búsqueda de funciones. * En el caso de VI, restringiremos el espacio donde buscaremos la distribución a posteriori. --- ## Inferencia Variacional (1) * Sea un modelo Bayesiano en el que todas las variables desconocidas tienen distribuciones a priori. * Denotamos con `\(Z\)` al conjunto de estas variables y de las variables latentes. * `\(X\)` es el conjunto de variables observadas. * El modelo probabilístico define `\(p(X, Z)\)`. * **Objetivo**: encontrar `\(p(Z \vert X)\)`, lo que requiere encontrar `\begin{equation} p(X) = \int p(X, Z) dZ \end{equation}` -- * Idea `\begin{equation} \arg \min_{q(Z)} KL(q(Z) \Vert p(Z \vert X)) \end{equation}` * `\(q(Z) = p(Z \vert X)\)`, no ganamos nada... --- ## Inferencia Variacional (2) * Alternativa: restringir espacio de búsqueda a familia de distribuciones `\(\mathcal{Q}\)` `\begin{equation} \arg\min_{q(Z) \in \mathcal{Q}} KL(q(Z) \Vert p(Z \vert X)) \end{equation}` * `\(\mathcal{Q}\)` ha de ser **suficientemente flexible** para ofrecer buenas aproximaciones, y al mismo tiempo **suficientemente simple** para ser tratable computacionalmente. --- ## Inferencia Variacional (3) * Sabemos que `\begin{eqnarray} KL[q(Z) \Vert p(Z \vert X)] &=& - \int q(Z) \log \left[ \frac{p(Z \vert X)}{q(Z)}\right] dZ \end{eqnarray}` * Que podemos escribir como `\begin{eqnarray} KL[q(Z) \Vert p(Z \vert X)] = \mathbb{E}[\log q(Z)] - \mathbb{E}[\log p(X,Z)] + \log p(X) \end{eqnarray}` * Evaluar la función objetivo requiere calcular `\(p(X)\)`!! -- * Minimizar la `\(KL\)` es equivalente a maximizar `\begin{eqnarray} ELBO(q) = \mathbb{E}[\log p(X,Z)] - \mathbb{E}[\log q(Z)] \end{eqnarray}` --- ## Inferencia Variacional (4) * Podemos escribir `\begin{eqnarray} ELBO(q) = \mathbb{E}[\log p(X\vert Z)] - KL[q(Z) \Vert p(Z)] \end{eqnarray}` * Maximizar el ELBO favorece a densidades que ponen masa en configuraciones que explican los datos (primer término), y al mismo tiempo a densidades cercanas a la distribución a priori (segundo término). * Además `\begin{eqnarray} \log p(X) = KL[q(Z) \Vert p(Z \vert X)] + ELBO(q) \end{eqnarray}` * Con lo que `\(\log p(X) \geq ELBO(q)\)`. * Esto explica que se haya usado `\(ELBO\)` para selección de modelos (como aproximador de la verosimilutud marginal). --- ## Aproximación de campo medio * Es una forma de restringir el espacio de búsqueda * Consiste en particionar los elementos de `\(Z\)` en grupos disjuntos `\(Z_i\)` con `\(i = 1, \dots, M\)`, y asumir `\begin{equation} q(Z) = \prod_{i=1}^M q_i(Z_i) \end{equation}` * Suele subestimar la varianza a posteriori... <center>  </center> --- ## Aproximación de campo medio. CAVI * Algoritmo para maximizar ELBO bajo la aproximación de campo medio. * Coordinate Ascent Variational Inference: actualiza cada factor de la densidad, manteniendo el resto de factores constante. * Converge a óptimo local. * Si fijamos todas las variables latentes menos `\(Z_j\)`, entonces el `\(q_j(Z_j)\)` óptimo es `\begin{equation} q^*_j(Z_j) \propto \exp \left[ \mathbb{E}_{-j} \log p(Z_j \vert Z_{-j}, X) \right] \end{equation}` * Que es lo mismo que `\begin{equation} q^*_j(Z_j) \propto \exp \left[ \mathbb{E}_{-j} \log p(Z_j , Z_{-j}, X) \right] \end{equation}` --- ## CAVI: Algoritmo 1. Inicializar los factors `\(q_i(Z_i)\)` 2. Actualizar cada factor utilizando `\begin{equation} q^*_j(Z_j) \propto \exp \left[ \mathbb{E}_{-j} \log p(Z_j \vert Z_{-j}, X) \right] \end{equation}` 3. Repetir paso 2 hasta convergencia. * Nota: cuando se da la **conjugación condicional**, entonces las condicionales completas de cada variable `\(p(Z_j \vert Z_{-j}, X)\)` se pueden escribir de forma analítica y la regla de actualización es muy sencilla. --- ## CAVI: Demostración * Podemos escribir el ELBO como `\begin{eqnarray} ELBO(q) &=& \mathbb{E}[\log p(X,Z)] - \mathbb{E}[\log q(Z)] \\ &=& \mathbb{E}_j \left[ \mathbb{E}_{-j} \left[ \log p(Z_j, Z_{-j}, X) \right] \right] -\mathbb{E}_j \left[\log q_j(Z_j) \right] + \text{cte} \end{eqnarray}` * Vemos que esta expresión es, salvo constante, menos la divergencia KL entre `\(q_j(Z_j)\)` y `\(q^*_j(Z_j)\)`. * Por tanto, maximizaremos el ELBO, escogiendo `\(q_j(Z_j) = q^*_j(Z_j)\)`. --- ## Inferencia Variacional Estocástica * CAVI no escala a datos masivos, pues en cada iteración requiere evaluar todo el dataset. * Alternativa: optimización estocástica del ELBO basada en el gradiente. * Requiere calcular una estimación ruidosa (insesgada) del gradiente del ELBO. * Para calcular esta estimación, no es necesario evaluar todos los datos. * <a href="https://arxiv.org/pdf/1601.00670.pdf">Variational Inference: A Review for Statisticians. </a> --- ## Automatic Differentiation Variational Inference * Otra posibilidad es **ADVI**. * Aproxima usando MC tanto el ELBO como su derivada para una forma paramétrica particular de la familia variacional (normal full-rank o mean field). * Requiere que sea sencillo muestrear de `\(q(Z)\)`. * Es la que usa STAN. * <a href="https://arxiv.org/pdf/1603.00788.pdf">Automatic Differentiation Variational Inference. </a>